|
Iniciación
a la bromatología (prácticas)
|
Protocolos
de análisis
|
Ref:
22.2
|
|
AMINOÁCIDOS
POR CG
|
||
OBJETIVO Y FUNDAMENTOS
Los trifluorobutilésteres de los aminoácidos pueden ser separados y cuantificados por cromatografía de gases. Estos trifluorobutilésteres aminoacídicos experimentan una retención selectiva dependiente de la temperatura, con etilenglicoladipato (EGA) o con metil-fenil-silicona ("rubber"), (OV-17)
La obtención de los
trifluorobutilésteres requiere:
1ro.- Hidrólisis de las proteínas.
2do.- Trifluorobutilación mediante fases intermedias: a)Conversión
en metilésteres b) Conversión de los metilésteres
a butilésteres. c) Obtención de trifluorobutilésteres.
Se utiliza ornitina como patrón
interno, por ser un aminoácido inexistente en las muestras objeto
del análisis.
Debe prepararse una mezcla estándar de ornitina y los aminoácidos
que interese determinar, simultáneamente con los problemas, a fin
de calcular los factores de respuesta y los tiempos de retención
relativos de cada aminoácido respecto a la ornitina.
MATERIAL
Balanza analítica.
Balanza granataria
Baló de 250 ml, esmerilado.
Columna de las características indicadas en la metodología.
Cromatógrafo de gases con registrador-integrador, equipamiento
complementario y detector de ionización de llama.
Cronómetro.
Reloj avisador.
Fluxómetro de burbuja.
Matraces aforados de 100 ml.
Microjeringa para C.G., graduada, de 10 µl.
Pipetas aforadas de 25 ml.
Placa calefactora.
Refrigerante de reflujo.
Tubos de ensayo 10 ml, con tapón roscado.
Evaporador rotatorio ("Buchi") con dispositivo de vacío.
Bomba de bacilo.
Baño termostático de agua.
Instalación protectora de la bomba de vacío.
Baño de parafina con agitador y termostato regulable.
Baterias de agitadores magnéticos.
Para muestras con alto contenido
azucares, es preciso, además:
Matrazs esmerilado forma balón de 1000 ml.
3 embudos de decantación esmerilados (en boca y en llave),
de 500 ml.
Columna con placa filtrante y llave, esmerilado hembra, de 2 cm
de F.
REACTIVOS
Ácido clorhídrico
1'25 N en alcohol metílico,obtenido según el procedimiento
de la práctica 22.1 .
Ácido clorhídrico 1'25 N en butanol, obtenido según
el procedimiento de la práctica 22.1, pero con butanol en lugar
de metanol.
Agua destilada.
Aire sintético calidad cromatográfica.
Hexano pa.
Hidrógeno calidad cromatográfica
Nitrógeno calidad cromatográfica.
Cloruro de metileno anhidro, grado espectrométrico.
Anhídrido trifluoroacético, calidad C.G.
Parafina líquida transparente incolora.
Gel de sílice (para desecación).
Cloruro cálcico (para desecación).
Ácido clorhídrico ~6N ( ½ HCl conc. y ½
H2O dest.).
HCl 0'1N pa.
Clorhidrato de ornitina patrón.
Disoluciones de aminoácidos patrón para test: En
un matraz aforado de 50 ml, disolver 130 mg de patrón de aminoácido
en 20-25 ml de HCl 0,1N (si se presentan dificultades para disolver algún
aminoácido, añadir unas gotas de HCl concentrado ), enrasar
a 50 ml con HCl 0'1N (una disolución para cada aminoácido).
Proceder igualmente con la ornitina.
Para muestras con contenido
alto de azúcares, es preciso, además:
Resina Dovex 50 WX-8 (40 cc).
Papel indicador.
Amoníaco 2N
NaOH diluido.
Nitrato de plata.
METODOLOGíA 1 (para muestras con contenido de azúcares bajo o moderado)
1.- Pesar una cantidad de
muestra, homogeneizada y molturada, que contenga unos 200-250 mg de proteína.
2.- Hidrolizar con HCl 6N a reflujo (~110 ºC) durante 22 horas.
3.- Dejar enfriar. Filtrar, lavando el residuo y recogiendo el
filtrado.
4.- Evaporar al vacío. a unos 60 ºC hasta sequedad
casi total. Redisolver con agua destilada y repetir la evaporación
2 veces más.
5.- Disolver en pequeñas cantidades de HCl 0'1 N y pasar
a un matraz aforado de 100 ml. Enrasar y homogeneizar.
6.- Pasar 25 ml a un matraz esmerilado de forma pera de 100 ml.
Añadir 2 ml del patrón interno de ornitina. Simultáneamente,
preparar una muestra estándar con 2 ml de patrón interno
y 2 ml de disolución patrón de cada uno de los aminoácidos
que queremos determinar. A partir de aquí, precederemos paralelamente
con esta muestra estándar.
7.- Evaporar al vacío. a 60 ºC hasta sequedad.
8.- Dejar un mínimo de 15 horas en un desecador al vacío.
9.- Añadir 10 ml de metanol/HCl 1'25N y agitar durante 30
minutos en placa magnética, a temperatura ambiente y con el frasco
tapado.
10.- Evaporar a 60 ºC hasta sequedad.
11.- Añadir 10 ml de n-butanol/HCl 1'25N, tapar con un tapón
de CaCl2 y calentar en un baño de parafina
a 100 ºC ± 1 ºC durante 2'5 horas y agitación
magnética.
12.- Evaporar al vacío. a 60 ºC hasta sequedad.
13.- Añadir 4 ml de diclorometano y 1 ml de anhídrido
trifluoracético.
14.- Colocar durante 15 minutos a temperatura ambiente y tapado.
15.- Pasar 2 ml a un tubo de presión con cierre hermético
a rosca y dejar 5 minutos a 150 ºC (ó 5 horas al ambiente).
16.- Enfriar el tubo con agua fria y guardar en frigorífico.
Sacar del frigorífico solo el tiempo necesario para inyectar en
el CG, a fin de que las condiciones del estándar y el problema
sean lo más idénticas que sea posible..
17.- Inyectar 2 µl en el CG.
METODOLOGÍA 2 (para muestras de elevado contenido de azúcares)
1.- Proceder como en los
puntos 1 al 3 del apartado anterior.
2.- Evaporar a casi sequedad al vacío. a 60 ºC.
3.- Diluir en agua destilada y llevar a pH aprox. de 3, mediante
solución de HCl o de NaOH. El volumen final no ha de sobrepasar
de los 300 ml.
4.- Pasar por una columna de 2 cm de F
rellena con 40 cc de resina Dowex WX-8, a un flujo máximo
de 0'5 ml/mn (se puede dejar a un flujo mucho más durante la noche),
según el proceso siguiente:
a) Activar la columna con 300 ml de HCl 6N.
b) Lavar con agua hasta test de cloruros negativo (prueba de turbidez
con nitrato de plata).
c) Pasar la muestra por la columna (retención de los aminoácidos).
d) Pasar agua destilada hasta pH neutro ( aproximadamente).
e) Pasar 250 ml de amoníaco 2N (elución de los aminoácidos)
f) Pasar 250 ml de agua (recoger estas dos últimas fracciones)
5.- Evaporar al vacío a 60 ºC hasta sequedad.
6.- Proceder siguiendo desde el punto 5 de la Metodología
1.
Parámetros cromatográficos
:
Gas portador = nitrógeno
Flujo de gas portador = 80 ml/mn.
Flujo de hidrógeno = 50 ml/mn
Flujo de aire = 450 ml/mn.
Temperatura inicial = 75 ºC
Temperatura final = 215 ºC.
Gradiente de temperatura ("rate") = 4ºC/mn
Detector = Ionización de llama.
Sensibilidad = 103 X 2.
Temperatura del detector = 350 ºC
Temperatura inyector = 200 ºC.
Columna = Vidrio, de 1'5 metros y 4 mm de diámetro interno,
con fase estacionaria de etilenglicoladipato al 0'6 % sobre soporte "Chromosorb
60-100".
CÁLCULOS
![]()
en donde:
%AX = % del aminoácido considerado,
sobre el total de la muestra.
AX = área del pico del aminoácido
considerado.
Asi = área del pico del patrón
interno (ornitina).
msi = masa de patrón interno (ornitina)
m = masa de muestra.
FC = factor de corrección del aminoácido.
El factor de corrección (FC) se calcula considerando:
![]()
en donde:
Fr = factor de respuesta relativo = F/Fe
siendo F el factor de respuesta absoluto del aminoácido
(Área/masa) i Fe el factor
de respuesta absoluto del estándar (Área del estándar
/ masa del estándar).
OBSERVACIONES
El tiempo de hidrólisis
de las muestras puede reducirse considerablemente, utilizando la presión
(en autoclave). Kaiser, Gehrke y otros investigadores han utilizado el
autoclave (4 horas a 145 ºC), obteniendo resultados tan aceptables
y reproducibles como los obtenidos por hidrólisis a 110 ºC.
Basándome en el método de los investigadores mencionados,
he comprobado empíricamente una relación entre el tiempo
de hidrólisis y la temperatura adecuada de hidrólisis, que
responde a la siguiente expresión:
![]()
en que T es la temperatura
en Kelvin y Ø el tiempo en horas, siendo a i b
unas constantes características para cada aminoácido;
para la lisina estos valores son bastante aproximadamente de a=1'300
i b=0'0488 (determinados empíricamente). Teóricamente,
cabe esperar ligeras desviaciones de los valores y la reproductibilidad
de a y b para la serina, tirosina, isoleucina, valina y leucina, desviaciones
bastante más apreciables para la metionina y francamente inadmisibles
para el triptófano.
El método no es válido para determinar cistina y
arginina (los picos no aparecen utilizando columna EGA) ni para el triptófano,
que queda destruido en buena parte durante la hidrólisis ácida.
La metionina queda parcialmente destruida durante la hidrólisis.
Si se desea determinar cistina y/o arginina, se utilizará
una columna de OV-17, pero hay que tener en cuenta que, por lo que respeta
a los otros aminoácidos. los resultados obtenidos con esa columna
son bastante menos fiables que los obtenidos con la columna EGA.
Para determinar triptófano, algunos autores han investigado
con hidrólisis alcalinas o con disoluciones de ácido tioglicólico.
El error por defecto para la metionina, puede compensarse multiplicando
el resultado obtenido por el factor empírico 1'25. Los resultados
son, sin embargo, poco reproducibles si los comparamos con otros aminoácidos,
pero por supuesto bastante mejores que los obtenidos mediante lo métodos
semicuantitativoss per cromatografía en capa fina.
Puede suprimirse el paso de la metilación efectuando la
butilacióncon n-butanol/HCl 3N y haciendo un tratamiento con ultrasonidos
durante 15 minutos a 100 ºC
Esquema de las reacciones químicas del proceso:
Metilación:
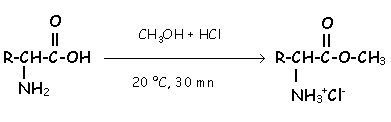
Butilación:
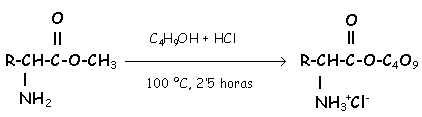
Trifluoroacetilación:
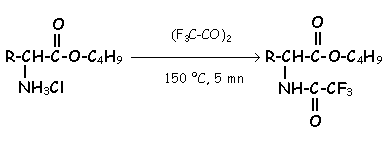
Aspecto de los cromatogramas
Los tiempos de retención relativos de los aminoácidos, referidos a la ornitina, con columnas EGA como la utilizada en esta práctica, son:
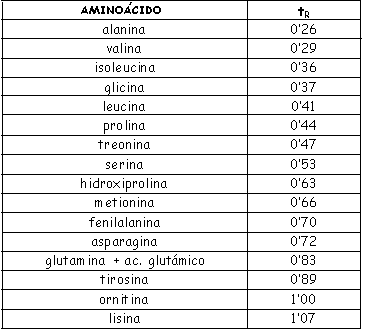
El ácido glutámico
y la glutamina aparecen como un único pico, por producirse una
transformación del ácido glutámico en glutamina durante
el proceso. El resultado se expresa como "ácido glutámico
+ glutamina expresados como glutamina".
El primer pico que aparece inmediatamente después de la
inyección corresponde al disolvente (diclorometano). A continuación
emerge un pico irregular y asimétrico que corresponde al exceso
de reactivo. A veces también pueden aparecer cierta cantidad de
pequeños picos correspondientes a productos de descomposición
y impurezas.
Cuestionario 22.2.- Aminoácidos por CG
1.- Deducir razonadamente las fórmulas utilizadas en los cálculos
2.- Hacer el esquema gráfico del procedimiento analítico
3.- Confeccionar el correspondiente "boletín de análisis".
4.- Idear un procedimiento para la determinación de aminoácidos por CG, basado en ese mismo, para el caso de que no dispusiésemos de patrón de ornitina.
5.- Diseñar un protocolo de análisis para poder trabajar simultáneamente con varias muestras.