- Primera etapa: Solució "Mare" de
PCR
Imatges
37 i 38: màquina de PCR
Per a preparar la PCR, s’agrega Solució "Mare" de
PCR a la mostra d’ADN (1) i (2). Aquesta
solució conté aigua, un buffer que manté la
barreja en al pH correcte, grans quantitats dels nucleòtids
d’adenina, timina, citosina i guanina, grans
quantitats de primers o encebadors d’ADN
que s’uneixen a la regió de l’ADN
que codifica al gen de la beta globina (per a iniciar
el procés de replicació), i ADN polimerasa
termoestable per a catalitzar l’allargament de
la cadena d’ADN.
Imatges
39 i 40: afegim els primers i col·loquem
els tubs a la màquina.
- Segona etapa: córrer
la PCR
Generalment la màquina posseeixun display que
indica tot allò que està succeint:
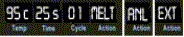
|
D’esquerra a dreta: temperatura, temps
restant, número del cicle, separació de
l’ADN (“Melt”), anellat (“ANL”) i
allargament (“EXT”) .
|
Imatge41: pantalla
de la màquina de PCR
El control es configura de la següent forma:
Període inicial = 10 minuts a 95°C
30 cicles de les següents seqüències
d’etapes:
- Separació de l’ADN (Melt):
30 segons a 95°C
- Anellat (Anneal): 30
segons a 60°C
- Allargament (Extend):
45 segons a 72°C
- Etapa final d’allargament:
10 minuts 72°C
- Etapa final: 4°C (conservar en
aquesta temperatura)
Imatges
42 i 43: fragment de la cadena
a seqüenciar i separació de la cadena
d’ADN.
Desnaturalització: Durant
cada cicle, la primer etapa (Melt)
està destinada a separar les cadenes de la doble
hèlix d’ADN, (5) i (6) ,per mitjà del
calentament a 95°C durant 30 segons.
Imatges
44 i 45: cadena d’ADN separada
i unió dels primers.
Anellament: Els primers o encebadors no
poden unir-se a l’ADN a aquesta temperatura,
per la qual cosa el tub es refreda a 60°C
(7). A aquesta temperatura els primers s’uneixen (anellen - Anneal)
a l’ADN monocatenari (8).
La raó per la qual les dues cadenes d’ADN
monocatenari no s’anellen entre sí, és
el gran excés de primers en la barreja,
per la qual cosa és molt més probable
que l’ADN monocatenari s’anelli amb un primer que
amb l’altra cadena d’ADN.
Imatges
46 i 47: cadena d’ADN amb
els primers i iniciació de d’activitat
de l’ADN polimerasa. Fi de l’activitat
de l’enzim.
Extensió: L’etapa final
(Extend) consisteix en pujar
la temperatura a 72°C durant 45 segons la qual
cosa permet a l’ADN polimerasa allargar la còpia
de la cadena d’ADN (9) y (10). Al final del cicle
obtenim ADN duplicat (11).
Imatges
48 i 49: obtenció de les
cadenes noves i fi de la PCR.
Les tres etapes juntes tarden menys
de 2 minuts en produir-se i ho fan en el mateix tub
on es troben. El cicle pot repetir-se fins a 30 vegades
més, i cada cadena nova d’ADN sintetitzada
actua com un nou motlle. Un cop fets els 30 o 35 cicles
es poden aconseguir fins a 1.000.000 de còpies
d’ADN.
· III - Separació de
l’ADN amplificat
Per a obtenir l’ADN amplificat
calseparar-lo de la resta en un gel d’agarosa.
A aquest gel en el que s’ha fet córrer l’ADN
se li retalla amb un “cuter” la banda corresponent
a l’ADN que ens interessa (en el cas que solament
hi hagi una banda no hi ha cap problema, en cas contrari,
cal saber quina és la banda corresponent a la
cadena que ens interessa).
· IV - Seqüenciació de
l’ADN
IVa. Seqüenciació de
l’ADN: PCR
Un cop que mostra està purificada
tot el producte de la PCR conté l’ADN
corresponent a la beta globina. Ara podem preparar
la mostra per a la seqüenciació automàtica.
En dos tubs amb una solució tampó, i
a una temperatura d’entre 40 i 50ºC, afegim
l’ADN amplificat. En un dels tubs s’afegeix
el primer que permet la polimerització de
l’ADN en una cadena, i en l’altre tub els
primers de la cadena complementària.
Imatges
50 i 51: preparació de la
seqüenciació.
Cada tub (1) conté doncs una
barreja seqüenciadora: buffers, primers (un
de diferent a cada tub), ADN polimerasa, nucleòtids,
acabadors fluorescents en proporcions adequades i es
fa córrer una altra PCR.
Imatges
52-59: activitat de l’enzim
i unió de les cadenes amb les substàncies
fluorescents.
L’objectiu en aquest cas no és
produir còpies idèntiques d’ADN,
sinó moltes còpies de diferents longituds(9).
Les figures (3,4,5,6,7,8,9 y 10) il·lustren
parcialment el que succeeix en un tub que conté el primer (..........
dret ). Cada cadena d’ADN s’uneix
al primer en un extrem i finalitza amb un "acabador" fluorescent
a l’altre extrem.
En la seqüenciació per
ciclació la primer etapa consisteix en crear
mitjançant el ciclador moltes còpies
de l’ADN "blanc", és a dir,
sense cap fluorescent. Tot i això, les còpies,
durant la replicació, s’acaben aleatòriament
en un punt determinar, per la qual cosa les còpies
són seqüències parcials de diferents
longituds.
La barreja de la reacció conté desoxinucleòtids
normals (A, C, G i T) i didesoxinucleòtids "especials" (A*,
C*, G*, i T*) que han estat marcats amb unes molècules
fluorescents. Els marcadors fluorescents són
diferents per a cada base nitrogenada, i cada un emet
una llum fluorescent amb un color diferent. Els didesoxinucleòtids
poden substituir a un desoxinucleòtid normal
durant la replicació però, si per casualitat
aquesta substitució ocorre, la cadena no pot
seguir allargant-se, acabant aquesta cadena d’ADN
en aquest punt, ja que no tenen extrems 3’ al
grup –OH que permet fer l’enllaç ester
amb el següent nucleòtid. . Els didesoxinucleòtids
són anomenats acabadors("terminators" ).
Mirem un exemple específic:
per a seqüenciar una cadena d’ADN (de cadena
simple) de 6 bases nitrogenades de longitud de la següent
seqüència:

Seguint l’exemple, amb aquest
mètode, es comencen a generar “trossos” des
de un fins a sis bases de longitud. Degut a que l’ADN
polimerasa estén la còpia d’ADN
des de l’extrem 5´ al 3´ (des del
final 3´ al final 5´ de l’ADN que
fa de motlle), aquestes peces serien, segons la correspondència
entre les bases:
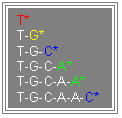
En tots aquests fragments la última
base (el final 3´) és un didesoxinucleòtid.
Una vegada que estan sintetitzats els fragments, ells
sols poden ser separats per la seva grandària:
una versió molt sensible de l’electroforèsi
en gel.
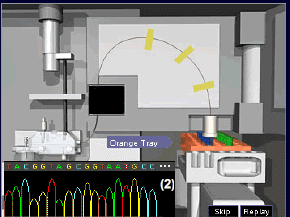
Imatge
60: inici de la seqüenciació de
l’ADN.
Donat que cada “tros” acaba
amb una base específica (per exemple T-G-C*termina
en C*), emetrà una llum fluorescent d’un
color específic, registrant el color dels fragments
d’ADN de mida creixent. Es pot deduir que la
seqüència de l’ADN complementari
(T-G-C-A-A-C), prové de la seqüència
original.
Per suposat, per a aquesta feina la
replicació sempre ha de començar al mateix
punt de l’ADN “blanc”. A l’exemple
anterior no ens interessaria trobar trossos com ara
G-C-A* (posició 2-3-4) interferint el procés....
Por altra banda, la mostra normal conté ADN
de doble cadena, la qual cosa significa que no solament
tenim el “motlle” (3' A-C-G-T-T-G 5'),
sinó que a més a més necessitem
la cadena complementària (5' T-G-C-A-A-C 3').
La cadena complementària pot originar trossos
com ara G-T-T*, laqual cosa no ens interessa. Aquests
problemes es solucionen utilitzant únicament primers que
anellen a seqüències específiques
(i conegudes) d’una sola de las cadenes.
A l’exemple següent, considerarem que en
comptes d’una cadena de sis bases, en tenim una
de deu bases, quatre més que les originals en
la direcció 3´.

Utilitzant com a primer la seqüència
3' A-C-A-T, la replicació sempre comença
al mateix lloc i les cadenes d’ADN formades són:

Donat que la incorporació dels acabadors succeeix
a l’atzar, resulta difícil fer una còpia
llarga si un desitja fer una còpia curta a la
mateixa reacció.
Per altra banda la separació de
les cadenes llargues per electroforèsi en base
a un nucleòtid de diferència és
bastant difícil. Per tant per a seqüenciar
una secció gran d’ADN s’utilitzen
molt primers diferents, cada un en el seu
tub corresponent, d’aquesta forma es seqüencien
en paral·lel diferents seccions parcialment
superposades (overlapping sections) i el resultat
es complica per a obtenir la seqüència
completa.
IVb Seqüenciació de
l’ADN: Electroforesi capil·lar
En la última etapa tenim 2
tubs que contenen el producte final de la PCR, una
barreja de fragments d’ADN de longituds diferents.
Tots els fragments d’ADN a cada tub comencen
amb el mateix primer però finalitzen amb un
nucleòtid diferent amb un marcador fluorescent
(amb un color diferent per a cada nucleòtid
A, T, G i C). La feina que queda per fer és
separar els fragments individuals d’ADN i identificar
el nucleòtid final.
Imatge
61 i 62: procés deseqüenciació de
l’ADN.
Això és realitza utilitzant
un seqüenciador automàtic que realitza
l’electroforèsi sobre gel de l’ADN
de cada tub. L’electroforèsi en gel és
un mètode que permet separar molècules
basades en diferències de mida. En aquest exemple
el seqüenciador posseeix un tub capil·lar
a l’extrem d’un mecanisme semblant a una
xeringa amb un buffer al seu interior. El tub s’acobla
al buffer i a l’altre extrem s’insereix
en un dels tubs que contenen les peces d’ADN.
S’aplica una corrent elèctrica de manera
que l’extrem del tub que estigui en contacte
amb l’ADN dugui la càrrega negativa i
la xeringa càrrega positiva.
Degut que l’ADN es troba carregat
negativament es mou al llarg del tub en direcció a
l’extrem, positiu (la xeringa). Els fragments
de mida més petita òbviament, es mouen
més ràpids que no pas els fragments més
grans. Prop de l’extrem del tub capil·lar
hi passa un raig làser que excita els marcadors
fluorescents, i uns detectors òptics determinen
el color de la fluorescència.
Podem assumir que en un conjunt de
fragments d’ADN (formats en l’etapa anterior)
tenim totes les mides diferents, que es diferencien
correlativament exactament en un nucleòtid.
La menor peça que conté una marca fluorescent
correspon al nucleòtid adjacent al primer. Aquest
fragment d’ADN correrà més ràpid
que els altres, llegint el color de la fluorescència,
així a mesura que passen per davant dels raigs
làser, es reconstrueix la seqüència
de bases de l’ADN (1) i (2).

Imatge
63: procés deseqüenciació de
l’ADN i muntatge de l’aparell de seqüenciació (esquerra)
i l’ordinador amb el programa que s’encarrega
de rebre la informació.